Подготовим файл координат и файл топологии. Используем предоставленный на прошлом задании gro файл с 38 молекулами этана для создания индекс-файла, в котором будет группа из одной молекулы этана:
make_ndx -f box_38.gro -o 1.ndxТеперь создадим gro файл с одной молекулой и зададим ячейку. При запуске editconf выберем номер, соответствующий группе из одной молекулы:
editconf -f box_38.gro -o et1.gro -n 1.ndx #зададим ячейку и расположим молекулу по центру ячейку editconf -f et1.gro -o et.gro -d 2 -cВ результате получаем файл et.gro. Исправим файл топологии et.top из прошлого задания (изменим в разделе [molecules] количество молекул этана с 38 на 1) и сохраним топологию в файле et1.top.
Даны 5 файлов с разными параметрами контроля температуры:
be.mdp - метод Берендсена для контроля температуры.
vr.mdp - метод "Velocity rescale" для контроля температуры.
nh.mdp - метод Нуза-Хувера для контроля температуры.
an.mdp - метод Андерсена для контроля температуры.
sd.mdp - метод стохастической молекулярной динамики.
Скрипт по работе с 5ю системами: script.bash
Сначала построим входные файлы для молекулярно-динамического движка mdrun с помощью grompp:
grompp -f ${i}.mdp -c et.gro -p et1.top -o et_${i}.tpr
# где i: be,vr,nh,an,sd список mdp файлов
Задавать i вне скрипта будем командой export i="be".
Для каждого из полученных 5 tpr файлов запускаем mdrun:
mdrun -deffnm et_${i} -v -nt 1
Теперь переходим к анализу результатов. Начнем с визуального анализа. Для этого проведем конвертацию в pdb.
trjconv -f et_${i}.trr -s et_${i}.tpr -o et_${i}.pdb
- Метод Берендсена: сначала молекулы слегка колеблются и вращаются, а потом амплитуда колебаний уменьшается и молекулы довольно быстро вращаются.
- Метод Андерсена: молекула не вращается, но видны небольшие колебания по длинам связей и валентным углам. Можно предположить, модель отражает систему этана в кристалле, то есть систему, в которой этан связан прочно.
- Метод Нуза-Хувера: молекула вращается, длина связи С-С в молекуле этана визуально практически не изменяется.
- Метод стохастической молекулярной динамики: молекула очень быстро перемещается, что сложно оценить, по какому принципу она движется.
- Метод "Velocity rescale": метод похож на метод Нуза-Хувера, однако вращение по связи меньше, а амплитуда колебаний больше.
Сравним потенциальную энергию связи и кинетическую энергию для каждой из 5 систем.
g_energy -f et_${i}.edr -o et_${i}_en.xvg
Построим графики изменения энергий в виде dot-plot.
set datafile commentschars "#@&" plot "./et_be_en.xvg" using 1:2, "./et_be_en.xvg" using 1:3 .... plot "./et_sd_en.xvg" using 1:2, "./et_sd_en.xvg" using 1:3
Метод Берендсена:
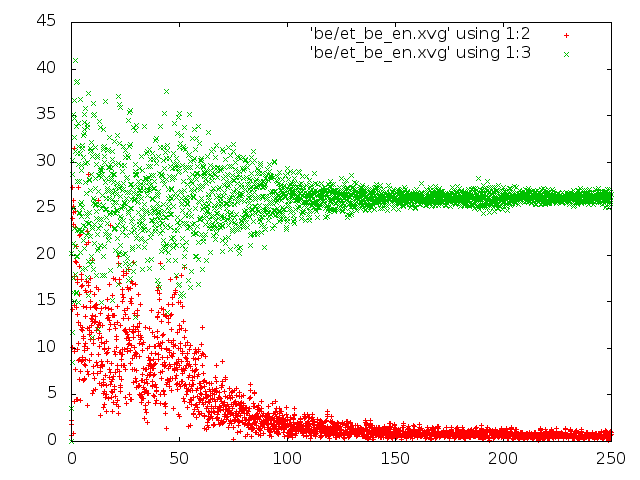
|
Метод Андерсена:
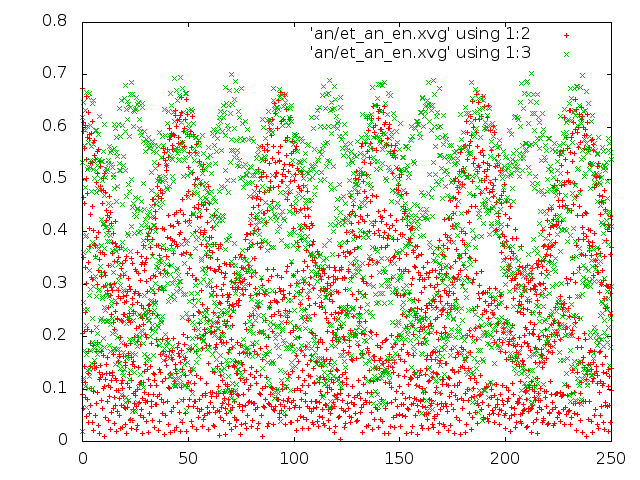
|
Метод Нуза-Хувера:
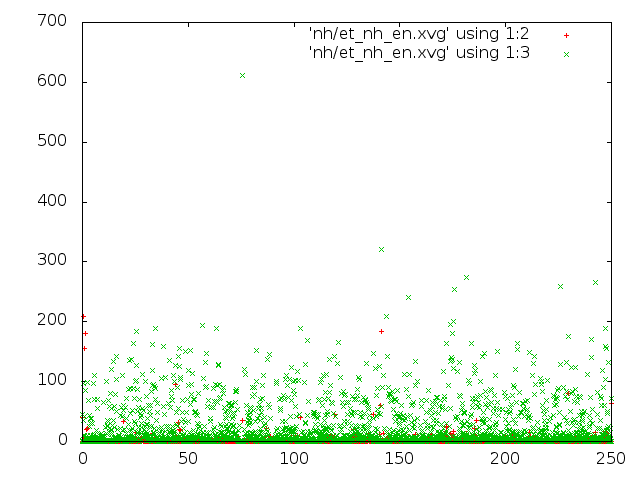
|
Метод стохастической молекулярной динамики:
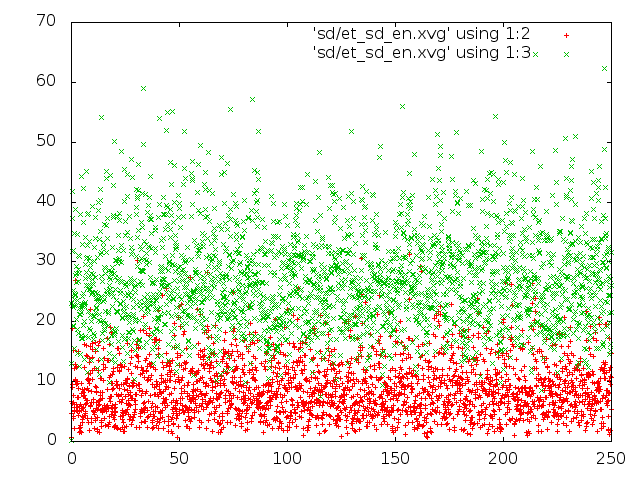
|
Метод "Velocity rescale":
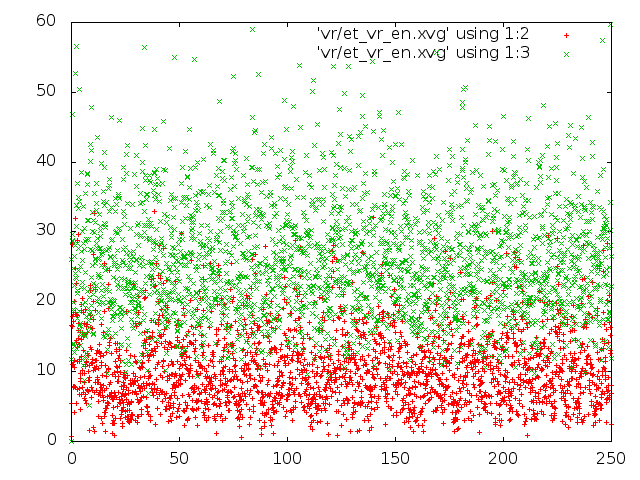
|
Рассмотрим распределение длинны связи С-С за время моделирования. Сначала создадим индекс файл с одной связью. Создадим файл b.ndx со следующим содержимым:
[ b ] 1 2И запустим утилиту по анализу связей g_bond:
g_bond -f et_${i}.trr -s et_${i}.tpr -o bond_${i}.xvg -n b.ndx
Построим графики распределения длинн связей в виде boxes в Gnuplot.
Метод Берендсена:
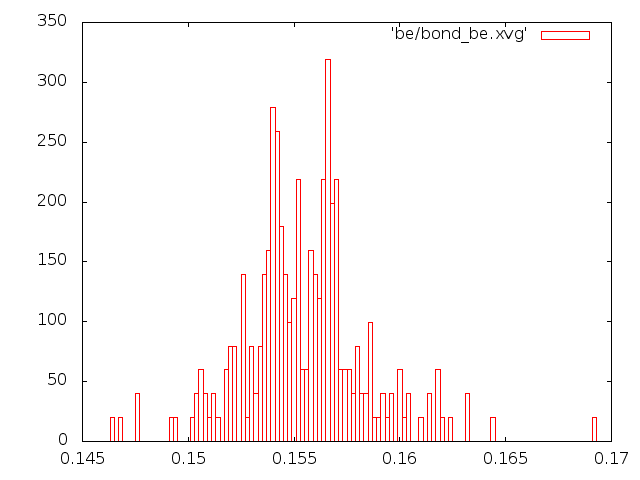
|
Метод Андерсена:
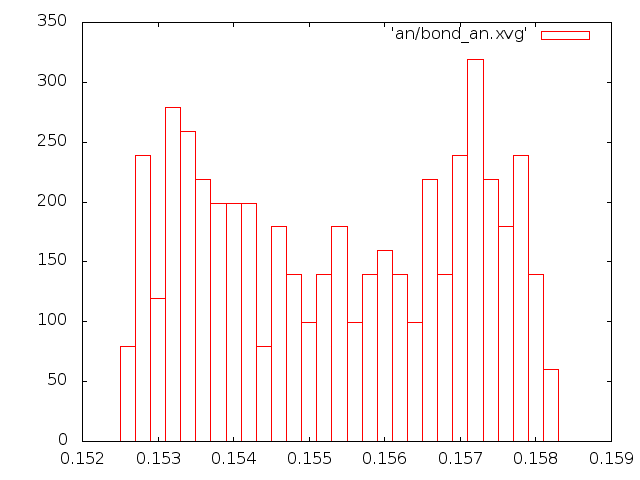
|
Метод Нуза-Хувера:
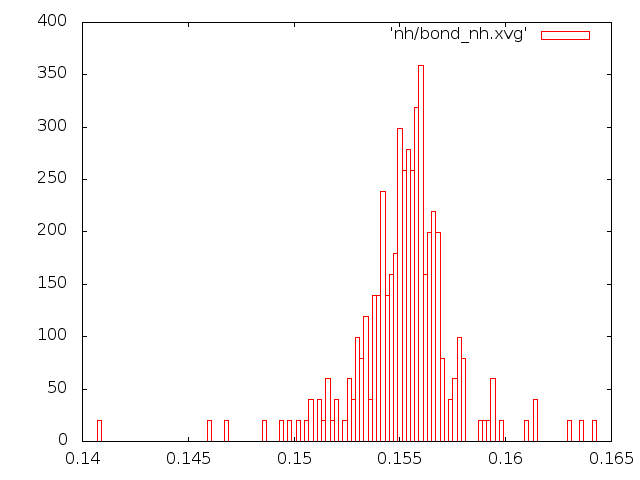
|
Метод стохастической молекулярной динамики:
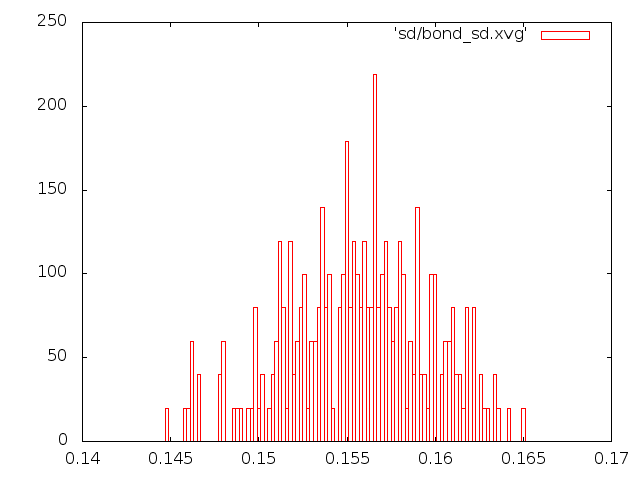
|
Метод "Velocity rescale":
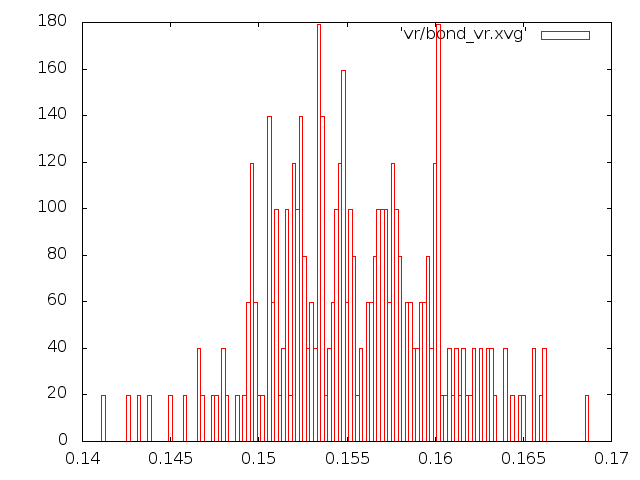
|
Сравним полученные наблюдения с распределением Максвелла-Больцмана:
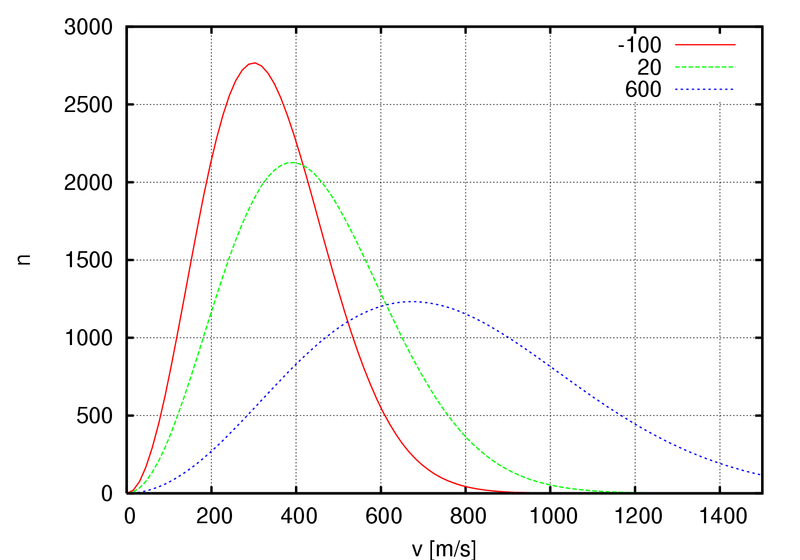
|
Такому распределению неплохо соответствуют графики методов "Velocity rescale", Нуза-Хувера и стохастической молекулярной динамики.
Наиболее реалисточно поддерживать температуру в системе позволяют, как мне кажется, методы "Velocity rescale" и Нуза-Хувера.