"Репрессор-активатор катаболизма", "Cra", "FruR", первоначальное название — "Репрессор фруктозы". Представляет собой бифункциональный транскрипционный регулятор: активатор и репрессор, играющий плейотропную роль в регулировании направления транспорта углерода в различных путях энергетического метаболизма, действует независимо от CRP-регулятора. По этой причине FruR регулятор вовлечен в экспрессию большого числа оперонов, продуктами которых явлются белки-ферменты центральных путей обмена углерода. FruR образует комплекс с ДНК в отсутствии фруктозы 1-фосфата или фруктозы 1,6-фосфата, связываясь с последовательностью в 18 нуклеотидов палиндромной последовательностью. В присутствии глюкозы повышается внутриклеточная концентрация фруктозы 1-фосфата и d-фруктозы 1,6-фосфата, которые взаимодействуют с FruR.
Go-термины: биологические процессы: гликолиз, транскрипция, ДНК-зависимая регуляция транскрипции, ответ на стимулы фруктозы; молекулярные функции: ДНК-связывание, транскрипция факторов активности, транскрипция активности активторов, транскрипция активности репрессоров; клеточный компонент: внутриклеточный, цитоплазмы
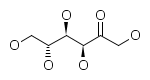
FruR принадлежит к семейству GalR-LacI транскрипционных регуляторов. Как представитель этого семейства белок содержит два функциональных домена: N-концевой домен, содержащий "спираль-поворот-спираль" мотив для связывания с ДНК; С-концевой домен связывания с эффектором. Изображение эффектора — d-фруктозы:
- Создание хорошего множественного выравнивания доменов заданной группы белков.
Построим множественное выравнивание ДНК-связывающих доменов группы специфичности laci.
Доменная организация белка-прототипа FruR_Ecoli:

Зеленым цветом обозначен ДНК-связывающий домен, красным - эффекторсвязывающий домен.
Все последовательности ДНК-связывающих доменов семейства LACI были получены из БД SMART через поиск по имени домена HTH_LACI. По нему было найдено представительское выравнивание доменов в формате FASTA: SMART.fasta и последовательности всех бактериальных доменов данного семейства в формате FASTA, которые были сохранены в файле: full.fasta
Для домена lacI было получено множественное выравнивание всех доменов этого типа. Чтобы получить из полного выравнивания выравнивание доменов заданной мне группе специфичности lacI, был написан скрипт: lacI.scr
Результат скрипта - файл dna_laci.fasta
При помощи программы ClustalW2 последовательности нужных ДНК-связывающих доменов были выровнены под профиль представительского выравнивания.
На выходе получился следующий файл: dna_laci_al_all.fasta
Из файла с выравниванием были вырезаны представительские последовательности. Полученное выравнивание было сохранено в файл dna_laci_al.fasta
- Создать единое множественное выравнивание заданных доменов всех групп специфичности
На этот раз для исследования мне была дана группа специфичности frur. Всё также необходимо исследовать ДНК-связывающий домен.
По аналогии с предыдущим заданием получим файл с множенственным выравниванием последовательностей нужных ДНК-связывающих доменов под профиль представительского выравнивания.
Выравнивание заданных для исследования доменов импортируем в Genedoc и объявим его последовательности новой группой (кнопка меню "Groups=>Edit sequence groups"). Назовем группу. Зададим также цвет для маркировки последовательностей этой группы. Затем последовательно будем добавлять по одному выравниванию доменов с разной специфичностью (кнопка "S"), каждый раз объявляя новую группу. Получим раскраску по группам (меню "Groups"), наиболее наглядно иллюстрирующую различия между группами.
В результате получим: файл с множенственным выравниванием, а также сдедующее изображение, где:
galrs - желтый gntr - голубой purr - фиолетовый scrr - голубой thur_rafr - черный trer - болотный laci - синий mali - розовый rbsr - оранжевый frur - сиреневый ptxs - серыйПозиции, консервативные для всего выравнивания: серин 19, лейцин-изолейцин-валин 22, валин-изолейцин 39, тирозин-фенилаланин 48.
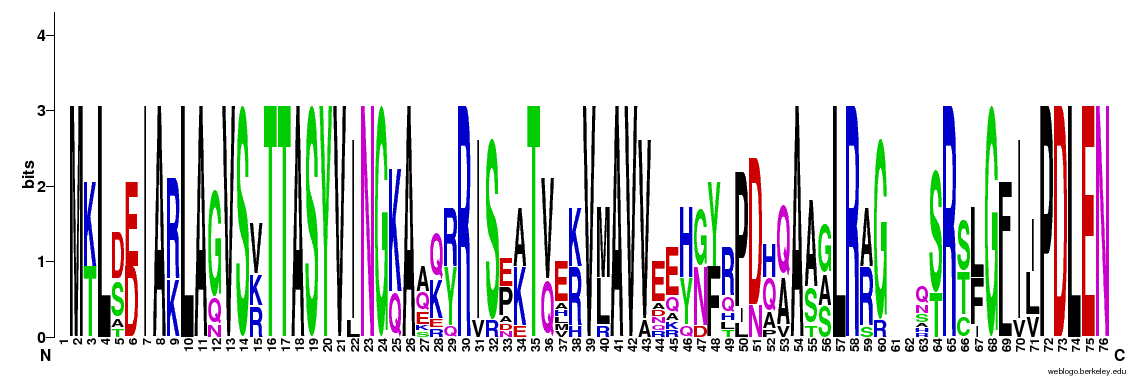
- Создание лого-изображения полного выравнивания заданных доменов и выравнивания доменов заданной группы специфичности. Используем ресурс WebLogo. ДНК-связывающий домен:
Logo для полного выравнивания:

Logo для выравнивания группы frur:
- Создание профиля для доменов белков группы специфичности frur Для начала надо добавить веса в выравнивании доменов белков. Для этого воспользуемся командой:
- Поиск белков заданной группы специфичности (frur) в протеоме Marinomonas sp. (strain MWYL1) Для этого скачаем файл с протеомом Marinomonas sp. (strain MWYL1). Затем проведем в протеоме поиск белков заданной группы специфичности (frur).
pfw -m frur.fasta > out.w.fasta
Выходной файл: out.w.fasta
Затем дадим на вход програме pfmake полученный файл для построения профиля:
pfmake -m out.w.fasta /usr/share/pftools23/blosum45.cmp > profile.prfВыходной файл: profile.prf
Затем дадим получившийся профиль программе autoscale для нормировки относительно случайной базы:
autoscale -m profile.prf > profile.scale.prf
Выходной файл: profile.scale.prf
Для этого воспользуемся программой pfsearch:
pfsearch -f profile.scale.prf proteom.fasta > pan.search;
Выходной файл: pan.search
pfsearch -x -f profile.scale.prf proteom > pan_s.fastaВыходной файл: pan_s.fasta
Далее используем программу ClustalW2 (на сервере kodomo-count запускается командой: /home/preps/golovin/progs/bin/clustalw2) для выравнивания найденных последовательностей под выравнивание семейства. На выходе получаем
выравнивание и изображение:
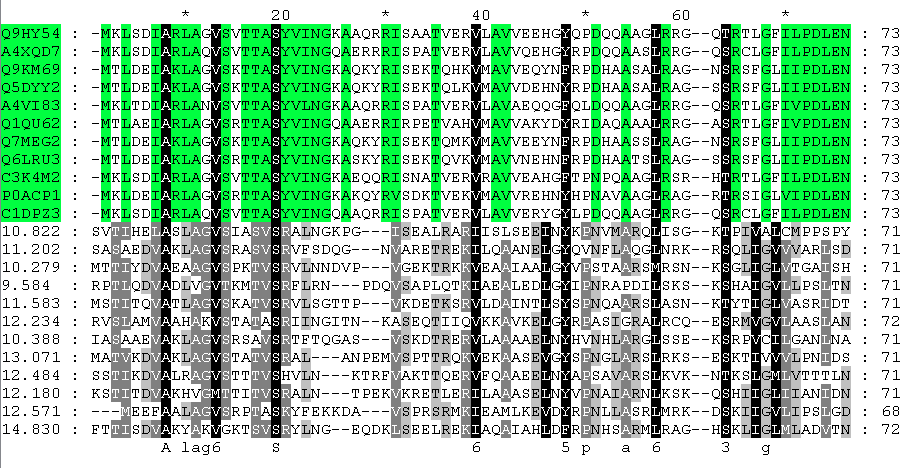
Зеленым выделены ДНК-связывающие домены группы специфичности frur.
Снизу расположены последовательности 12 белковых находок.
Данные находки вряд ли принадлежат к группе специфичности frur, т.к. они, как минимум, не содержат специфических позиций, характерных для данной группы специфичности, т.е. уже на этом этапе они не проходят отбор.