Программы DSSP и HBplus
- Определить вторичную структуру молекул белка, представленных в записи 1jw9, программой DSSP. Перечислить цепи белка и для каждой – сколько каких элементов вторичной структуры нашёл DSSP. Одним элементом вторичной структуры считаем блок идущих подряд букв H (альфа-спираль) или E (бета-тяж). Открыть PDB-файл в редакторе и изучить поля HELIX и SHEET. Сравнить два описания вторичной структуры (вычисленное DSSP и приведённое авторами записи).
Имея pdb-файл записи 1jw9 (1jw9.pdb), запустив на kodomo команду:
dsspcmbi 1jw9.pdb 1jw9.dsspполучим на выходе dssp (1jw9.dssp).
Выходной файл импортирую в excel (dssp.xls). Необходимая нам информация заключена всего в двух колонках: RESIDUE (где приведены номер остатка и
идентификатор цепи) и STRUCTURE, из содержимого каждой из ячеек которой нам нужен только первый символ. Используя данные выдачи dssp, получу информацию
об элементах вторичной структуры для каждой цепи.Цепь B:
1 альфа-спираль: 6-11
2 альфа спираль: 13-16
3 альфа спираль: 22-31
4 альфа спираль: 41-53
5 альфа спираль: 86-97
6 альфа спираль: 112-120
7 альфа спираль: 132-145
8 альфа спираль: 174-177
9 альфа спираль: 194-2131 бета-тяж: 33-37
2 бета-тяж: 57-61
3 бета-тяж: 102-106
4 бета-тяж: 124-127
5 бета-тяж: 149-155
6 бета-тяж: 158-164
7 бета-тяж: 222-227
8 бета-тяж: 232-237Цепь D:
1 альфа-спираль: 8-14
2 альфа спираль: 28-36
3 альфа спираль: 40-451 бета-тяж: 2-6
2 бета-тяж: 18-21
3 бета-тяж: 52-55
4 бета-тяж: 58-59
5 бета-тяж: 71-75Теперь посмотрим данные о вторичной структуре, приведенные авторами в pdb-файле.
Цепь B:
1 альфа-спираль: 5-12
2 альфа спираль: 12-17
3 альфа спираль: 21-32
4 альфа спираль: 40-54
5 альфа спираль: 67-73
6 альфа спираль: 78-82
7 альфа спираль: 85-98
8 альфа спираль: 111-121
9 альфа спираль: 131-146
10 альфа спираль: 172-178
11 альфа спираль: 193-2141 бета-тяж: 33-37
2 бета-тяж: 57-61
3 бета-тяж: 102-106
4 бета-тяж: 124-127
5 бета-тяж: 149-155
6 бета-тяж: 158-164
7 бета-тяж: 222-227
8 бета-тяж: 232-237Цепь D:
1 альфа-спираль: 7-15
2 альфа спираль: 27-37
3 альфа спираль: 39-461 бета-тяж: 2-6
2 бета-тяж: 18-21
3 бета-тяж: 52-55
4 бета-тяж: 58-59
5 бета-тяж: 71-75Теперь сравним результаты. DSSP не нашел две альфа-спирали цепи B, которые числятся в pdb (выделены жирным выше - 67-73 и 78-82). В выдаче dssp эти две последовательности остатков считаются G = 3-helix (3/10 helix) . Все остальные элементы вторичной структуры полностью совпали, если учесть, что в dssp в альфа-спираль не включены первый и последний ее остатки (таков алгоритм программы).
2. Проанализировать колонку PHI выдачи DSSP. Перечислить остатки, имеющие положительное значение угла φ (указать тип остатка, номер и цепь). Создать в PyMOL изображение, представляющее остовную модель (ribbon) одной из цепей белка, на котором различными цветами были бы выделены альфа-спирали, бета-тяжи, остатки с положительным φ и все прочие.
Из файла-выдачи dssp выпишу остатки с положительным значением угла φ:
аланин 2 цепи B;
аспарагиновая кислота 22 цепи B;
глицин 38 цепи B;
глицин 54 цепи B;
глицин 83 цепи B;
лизин 146 цепи B; аргинин 156 цепи B;
тирозин 166 цепи B;
глицин 169 цепи B;
глутаминовая кислота 189 цепи B; глицин 214 цепи B;
глицин 216 цепи B;
глицин 221 цепи B;
цистеин 231 цепи B;
метионин 1 цепи D; аланин 8 цепи D;
глицин 15 цепи D;
глицин 49 цепи D; аспарагин 56 цепи D; глутамин 57 цепи D;
глицин 69 цепи D;
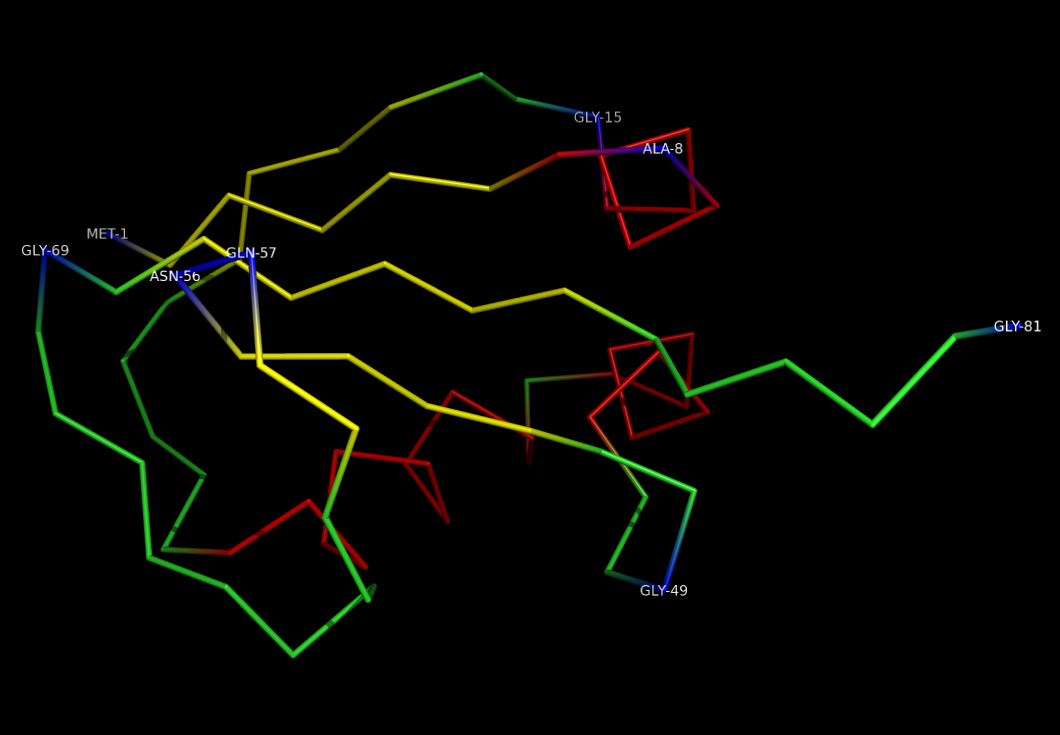
глицин 81 цепи D;Создам в PyMOL изображение, представляющее остовную модель (ribbon) цепи D белка, на котором различными цветами выделены альфа-спирали, бета-тяжи, остатки с положительным значением угла φ и все прочие:
Красным изображены альфа-спирали, желтым - бета-тяжи, синим - остатки с положительным значением угла φ, зеленым - все остальные остатки.
3. Программой HBPlus определить водородные связи в данной записи. Привести примеры водородных связей а) участвующих в стабилизации вторичной структуры; б) между боковыми цепями аминокислотных остатков; в) между боковой цепью одного остатка и остовным атомом другого. Есть ли в структуре водородные связи между белком и каким-либо лигандом (малой органической молекулой)? Если есть, то привести пример. Привести также один-два примера водяных мостиков между различными остатками. Каждый пример должен быть проиллюстрирован изображением, сгенерированным в PyMOL.
На kodomo командой:
hbplus 1jw9.pdbполучим файл 1jw9.hb2 с водородными связями в данной записи.
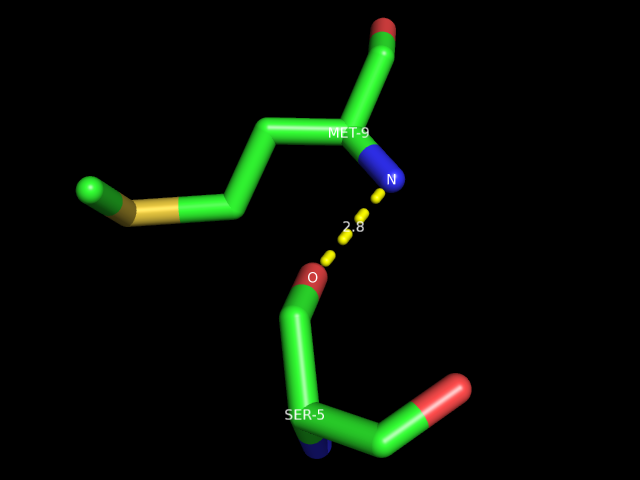
а. водородная связь, учавствующая в стабилизации вторичной структуры. Например, водородная связь между атомом кислорода остатка Ser-5 цепи B и атомом азота остатка Met-9 цепи B. Эти остатки входят в альфа-спираль и иллюстрируют правило, согласно которому остовный атом кислорода n-го остатка образует водородную связь с остовным азотом (n+4)-го остатка:
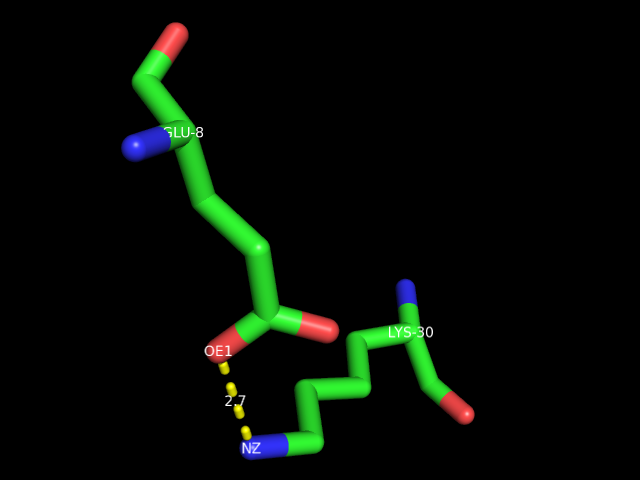
б. водородная связь между боковыми цепями аминокислотных остатков. Например, водородная связь между атомом азота аминогруппы боковой цепи остатка Lys-30 цепи B и атомом кислорода карбоксильной группы боковой цепи остатка GLU-8 цепи B:
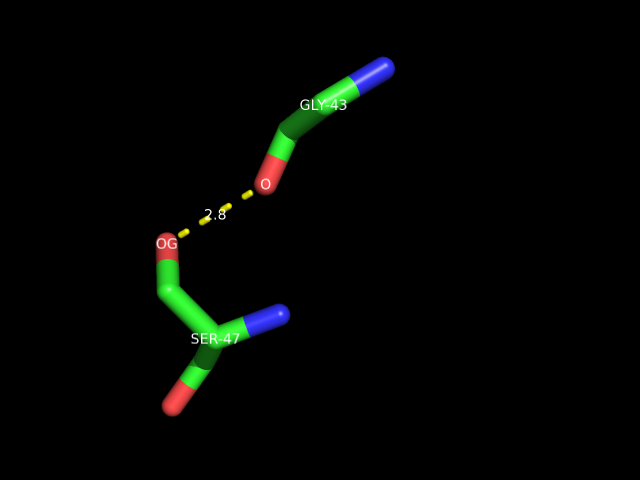
в. водородная связь между боковой цепью одного остатка и остовным атомом другого. Например, водородная связь между атомом кислорода гидрокисльной группы боковой цепи остатка SER-47 цепи B и остовным атомом кислорода остатка GLY-43 цепи B:
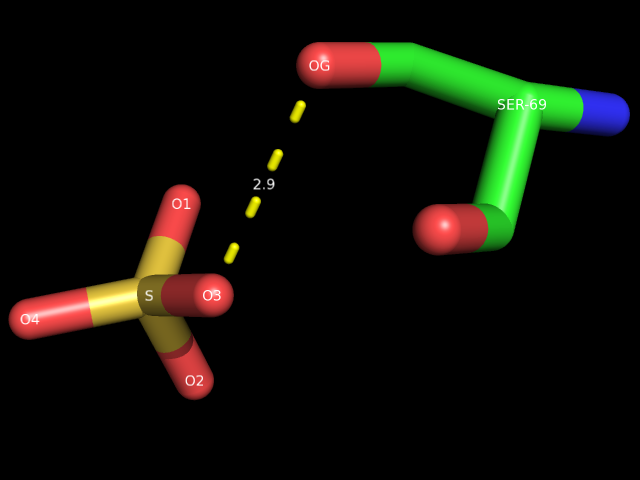
г. водородная связь между белком и лигандом. Например, водородная мвязь между атомом кислорода гидрокисльной группы боковой цепи остатка SER-69 (цепь B) и атомом кислорода лиганда SO4 (252 цепь B):
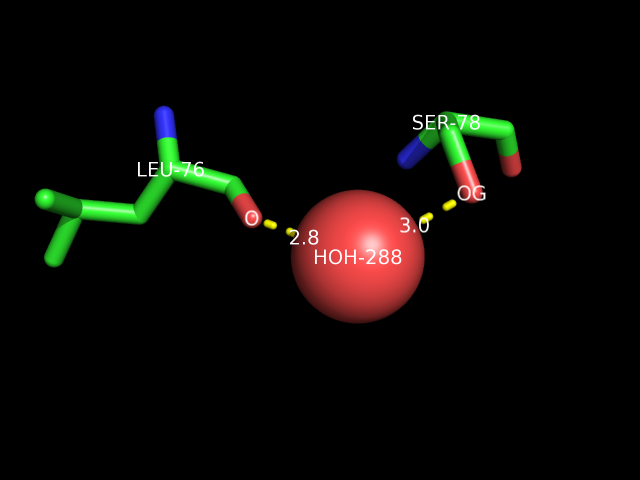
д. водяной мостик между различными остатками. Например, водяной мостик между атомом кислорода гидрокисльной группы боковой цепи остатка SER-78 (цепь B) и атомом кислорода остатка LEU-76 цепи B посредством водородных связей с молекулой воды 288:
©2008-2010 Михальченко Алексей