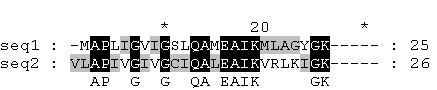Программа BLASTP
- Поиск гипотетических гомологов изучаемого белка в разных БД
Таблица 1. Результаты поиска гипотетических гомологов белка MOEB_ECOLI
Поиск по БД Swiss-Prot Поиск по БД PDB Поиск по БД "nr" 1. Лучшая находка Идентификатор БД MOEB_ECOLI 1JWB цепь B NP_415347.1 E-value 5e-144 3e-145 8e-143 Вес (в битах) 509 509 509 % идентичности 100% 100% 100% Найдены ли другие белки с теми же значениями E-value и веса в битах?
Если найдены, то укажите общее число и приведите один идентификатор (любой, но желательно Swiss-Prot ID)не найдены не найдены не найдены 2. Сколько хороших кандидатов в гомологи найдено? (число находок в списке описаний, Descriptions, с E-value < 1E-10) 37 6 1896 2. "Худшая" находка (последняя в выдаче с E-value < 1) Номер находки в списке описаний (Descriptions) 120 21 3486 Идентификатор БД PDXB_SHELP 2GAG цепь B YP 002603283.1 E-value 0.91 0.71 0.98 Вес (в битах) 33.9 30.0 37.7 % идентичности 31% 32% 25% % сходства 43% 53% 43% Длина выравнивания 112 56 81 Координаты выравнивания (номера первых и последних а.о.) 26-137 в MOEB_ECOLI
112-216 в PDXB_SHELP21-75 в MOEB_ECOLI
11-66 в 2GAG цепь B50-130 в MOEB_ECOLI
62-142 в YP 002603283.1% гэпов 6% 1% 0%
- изучаемый белок удалось найти в Swiss-Prot , а его структуру в PDB, в "nr" белка не оказалось;
- параметры выравнивания изучаемого белка с самим собой при поиске по разным БД совпадают за исключением E-value, так как при расчете E-value учитывается объем банка данных. Так, меньшим E-value получился в банке PDB (из рассмотренных БД он содержит меньше всего записей) - 3е-145, а в Swiss-Prot большим (этот банк содержит примерно в десять раз больше записей, чем PDB) - 5е-144. В "nr" изучаемого белка не оказалось;
- число потенциальных гомологов при поиске по разным БД меньшим оказалось в PDB и большим в "nr", что связано с объемами банков (чем больше банк, тем больше вероятность найти потенциальных гомологов);
- "худшие" находки при поиске по разным БД не совпадают. Это связано с разными объемами БД. Если выбирать из "худших" находок самую хорошую только сравнивая E-value из таблицы выше, то таковой будет находка из PDB (0,71 < 0,91 < 0,98). Но если учитывать, что находка из "nr" в двух остальных банках имела бы намного меньший E-value (т.е. была бы лучшей), то самой хорошей из "худших" находок можно считать находку из "nr";
- Поиск гипотетических гомологов изучаемого белка с фильтром по таксонам
Задача — для изучаемого белка E. coli найти лучшего гомолога в организмах таксона, филогенетически как можно более далекого от E. coli.
Для исследования предлагаются следующие таксоны: Homo sapiens, Archaea, Actinobacteria, Alteromonadales, Vibrionaceae (приведены в порядке приближения к E. coli). В этом же порядке проверяйте на наличие гипотетического гомолога (критерий: E-value<0,001).В организмах таксона Homo sapiens лучший гомолог - MOCS3_HUMAN (Molybdenum cofactor synthesis protein 3). Его E-value 1e-46, вес в битах 182, идентичность - 45%, сходство - 60%, гэпов - 1%, координаты выравнивания: 2-233 в MOEB_ECOLI, 53-285 в MOCS3_HUMAN, длина выравнивания - 234.
- Поиск белка по его фрагменту
VLAPIVGIVGCIQALEAIKVRLKIGK - фрагмент белка.
>sp|P45211|MOEB_HAEIN Molybdopterin biosynthesis protein moeB
MIELSHEEELRYNRQIILKSVDFDGQEKLKASKMLIVGLGGLGCAASQYLAAAGVGNLTLLDFDTVSLSN
LQRQVLHCDARLNMPKVESAKIALEQINPHINIETINAKLDEEKLAEIIPHFDIVLDCTDNVEIRNQLDR
QCNHMKVPLISGAAIRMEGQVSVFTYEPNTPTYRDLSKLFRQNVLSCVEAGVLAPIVGIVGCIQALEAIK
VRLKIGKNLCGRLLMIDGFSMNIREIKLPTNMEТаблица 2. Результаты поиска белка в Swiss-Prot по фрагменту последовательности
Поиск по фрагменту Поиск по полной
последовательностиАС лучшей находки P45211 P45211 E-value 1e-16 5e-140 Вес (в битах) 82.9 496 Найдены ли другие белки с теми же значениями E-value и веса в битах? не найдены не найдены Лучшей находкой является один и тот же белок, только E-value и вес во втором случае намного лучше, так как длина полной последовательности больше длины фрагмента, а значит и совпадение полной последовательности с полной последовательностью лучше, чем фрагмента с полной последовательностью.
На втором месте находится мой белок MOEB_ECOLI. Выравнивание:
MOEB_HAEIN 1 MIELSHEEELRYNRQIILKSVDFDGQEKLKASKMLIVGLGGLGCAASQYLAAAGVGNLTL 60
M ELS +E LRYNRQIIL+ DFDGQE LK S++LIVGLGGLGCAASQYLA+AGVGNLTL
MOEB_ECOLI 1 MAELSDQEMLRYNRQIILRGFDFDGQEALKDSRVLIVGLGGLGCAASQYLASAGVGNLTL 60MOEB_HAEIN 61 LDFDTVSLSNLQRQVLHCDARLNMPKVESAKIALEQINPHINIETINAKLDEEKLAEIIP 120
LDFDTVSLSNLQRQ LH DA + PKVESA+ AL +INPHI I +NA LD+ +LA +I
MOEB_ECOLI 61 LDFDTVSLSNLQRQTLHSDATVGQPKVESARDALTRINPHIAITPVNALLDDAELAALIA 120MOEB_HAEIN 121 HFDIVLDCTDNVEIRNQLDRQCNHMKVPLISGAAIRMEGQVSVFTYEPNTPTYRDLSKLF 180
D+VLDCTDNV +RNQL+ C KVPL+SGAAIRMEGQ++VFTY+ P YR LS+LF
MOEB_ECOLI 121 EHDLVLDCTDNVAVRNQLNAGCFAAKVPLVSGAAIRMEGQITVFTYQDGEPCYRCLSRLF 180MOEB_HAEIN 181 RQNVLSCVEAGVLAPIVGIVGCIQALEAIKVRLKIGKNLCGRLLMIDGFSMNIREIKLPT 240
+N L+CVEAGV+AP++G++G +QA+EAIK+ GK G+++M D + RE+KL
MOEB_ECOLI 181 GENALTCVEAGVMAPLIGVIGSLQAMEAIKMLAGYGKPASGKIVMYDAMTCQFREMKLMR 240MOEB_HAEIN 241 N 241
N
MOEB_ECOLI 241 N 241 (жирным шрифтом указан фрагмент выравнивания в GeneDoc)Вот выравнивание, сделанное мной в GeneDoc:
Если учитывать, что "V", стоящий напротив "V" в полном выравнивании, не входил в короткую последовательность, то выравнивания совпали.
- Сравнение выравниваний, выданных программой BLASTP, с оптимальными глобальным и локальным выравниваниями.
C помощью програм needle и water получим получу оптимальное глобальное и локальное выравнивания белков MOEB_HAEIN и MOEB_ECOLI, установив штрафы такие же как в BLASTP (за открытие пропуска - 11, за продолжение - 1).
Выравнивание BLASTP:
Score = 319 bits (817),
Expect = 1e-86,
Identities = 154/241 (63%)
Positives = 189/241 (78%),
Gaps = 0/241 (0%)
MOEB_HAEIN 1 MIELSHEEELRYNRQIILKSVDFDGQEKLKASKMLIVGLGGLGCAASQYLAAAGVGNLTL 60
M ELS +E LRYNRQIIL+ DFDGQE LK S++LIVGLGGLGCAASQYLA+AGVGNLTL
MOEB_ECOLI 1 MAELSDQEMLRYNRQIILRGFDFDGQEALKDSRVLIVGLGGLGCAASQYLASAGVGNLTL 60MOEB_HAEIN 61 LDFDTVSLSNLQRQVLHCDARLNMPKVESAKIALEQINPHINIETINAKLDEEKLAEIIP 120
LDFDTVSLSNLQRQ LH DA + PKVESA+ AL +INPHI I +NA LD+ +LA +I
MOEB_ECOLI 61 LDFDTVSLSNLQRQTLHSDATVGQPKVESARDALTRINPHIAITPVNALLDDAELAALIA 120MOEB_HAEIN 121 HFDIVLDCTDNVEIRNQLDRQCNHMKVPLISGAAIRMEGQVSVFTYEPNTPTYRDLSKLF 180
D+VLDCTDNV +RNQL+ C KVPL+SGAAIRMEGQ++VFTY+ P YR LS+LF
MOEB_ECOLI 121 EHDLVLDCTDNVAVRNQLNAGCFAAKVPLVSGAAIRMEGQITVFTYQDGEPCYRCLSRLF 180MOEB_HAEIN 181 RQNVLSCVEAGVLAPIVGIVGCIQALEAIKVRLKIGKNLCGRLLMIDGFSMNIREIKLPT 240
+N L+CVEAGV+AP++G++G +QA+EAIK+ GK G+++M D + RE+KL
MOEB_ECOLI 181 GENALTCVEAGVMAPLIGVIGSLQAMEAIKMLAGYGKPASGKIVMYDAMTCQFREMKLMR 240MOEB_HAEIN 241 N 241
N
MOEB_ECOLI 241 N 241
Оптимальное глобальное выравнивание:
# Aligned_sequences: 2 # 1: MOEB_HAEIN # 2: MOEB_ECOLI # Matrix: EBLOSUM62 # Gap_penalty: 11.0 # Extend_penalty: 1.0 # Length: 249 # Identity: 154/249 (61.8%) # Similarity: 189/249 (75.9%) # Gaps: 6/249 ( 2.4%) # Score: 784.0MOEB_HAEIN 1 MIELSHEEELRYNRQIILKSVDFDGQEKLKASKMLIVGLGGLGCAASQYL 50 |.|||.:|.|||||||||:..||||||.||.|::|||||||||||||||| MOEB_ECOLI 1 MAELSDQEMLRYNRQIILRGFDFDGQEALKDSRVLIVGLGGLGCAASQYL 50MOEB_HAEIN 51 AAAGVGNLTLLDFDTVSLSNLQRQVLHCDARLNMPKVESAKIALEQINPH 100 |:||||||||||||||||||||||.||.||.:..||||||:.||.:|||| MOEB_ECOLI 51 ASAGVGNLTLLDFDTVSLSNLQRQTLHSDATVGQPKVESARDALTRINPH 100MOEB_HAEIN 101 INIETINAKLDEEKLAEIIPHFDIVLDCTDNVEIRNQLDRQCNHMKVPLI 150 |.|..:||.||:.:||.:|...|:||||||||.:||||:..|...||||: MOEB_ECOLI 101 IAITPVNALLDDAELAALIAEHDLVLDCTDNVAVRNQLNAGCFAAKVPLV 150MOEB_HAEIN 151 SGAAIRMEGQVSVFTYEPNTPTYRDLSKLFRQNVLSCVEAGVLAPIVGIV 200 ||||||||||::||||:...|.||.||:||.:|.|:||||||:||::|:: MOEB_ECOLI 151 SGAAIRMEGQITVFTYQDGEPCYRCLSRLFGENALTCVEAGVMAPLIGVI 200MOEB_HAEIN 201 GCIQALEAIKVRLKIGKNLCGRLLMIDGFSMNIREIKLPTNME------ 243 |.:||:||||:....||...|:::|.|..:...||:||..|.. MOEB_ECOLI 201 GSLQAMEAIKMLAGYGKPASGKIVMYDAMTCQFREMKLMRNPGCEVCGQ 249Длина выравнивания в needle больше длины выравнивания BLASTP. В нем присутстуют на конце 8 колонок, которые не представлены в выравнивании BLASTP. В позициях с 1 по 241 выравнивания полностью совпадают.
Оптимальное локальное выравнивание:
# Aligned_sequences: 2 # 1: MOEB_HAEIN # 2: MOEB_ECOLI # Matrix: EBLOSUM62 # Gap_penalty: 11.0 # Extend_penalty: 1.0 # Length: 241 # Identity: 154/241 (63.9%) # Similarity: 189/241 (78.4%) # Gaps: 0/241 ( 0.0%) # Score: 788.0MOEB_HAEIN 1 MIELSHEEELRYNRQIILKSVDFDGQEKLKASKMLIVGLGGLGCAASQYL 50 |.|||.:|.|||||||||:..||||||.||.|::|||||||||||||||| MOEB_ECOLI 1 MAELSDQEMLRYNRQIILRGFDFDGQEALKDSRVLIVGLGGLGCAASQYL 50MOEB_HAEIN 51 AAAGVGNLTLLDFDTVSLSNLQRQVLHCDARLNMPKVESAKIALEQINPH 100 |:||||||||||||||||||||||.||.||.:..||||||:.||.:|||| MOEB_ECOLI 51 ASAGVGNLTLLDFDTVSLSNLQRQTLHSDATVGQPKVESARDALTRINPH 100MOEB_HAEIN 101 INIETINAKLDEEKLAEIIPHFDIVLDCTDNVEIRNQLDRQCNHMKVPLI 150 |.|..:||.||:.:||.:|...|:||||||||.:||||:..|...||||: MOEB_ECOLI 101 IAITPVNALLDDAELAALIAEHDLVLDCTDNVAVRNQLNAGCFAAKVPLV 150MOEB_HAEIN 151 SGAAIRMEGQVSVFTYEPNTPTYRDLSKLFRQNVLSCVEAGVLAPIVGIV 200 ||||||||||::||||:...|.||.||:||.:|.|:||||||:||::|:: MOEB_ECOLI 151 SGAAIRMEGQITVFTYQDGEPCYRCLSRLFGENALTCVEAGVMAPLIGVI 200MOEB_HAEIN 201 GCIQALEAIKVRLKIGKNLCGRLLMIDGFSMNIREIKLPTN 241 |.:||:||||:....||...|:::|.|..:...||:||..| MOEB_ECOLI 201 GSLQAMEAIKMLAGYGKPASGKIVMYDAMTCQFREMKLMRN 241Внешне выравнивыние BLASTP не отличается от оптимального локального выравнивания.
Сравним параметры трех выравниваний:
Сравним полученные значения выравниваний:
Параметр BLASTP needle water Длина 241 249 241 Идентичность 154/241 (63%) 154/249 (61.8%) 154/241 (63.9%) Сходство 189/241 (78%) 189/249 (75.9%) 189/241 (78.4%) Гэпы 0/241 (0%) 6/249 ( 2.4%) 0/241 ( 0.0%) Вес 817 784 788
Параметры выравнивания BLAST и выравнивание в water немного отличаются, так как BLASTP округляет значения идентичности и сходства. При приблизительно равных параметрах этих двух выравниваний вес их отличается довольно сильно. Так как гэпов ни там, ни там не обнаружено (в обратном случае различие можно было бы объяснить лишним учетом штрафа за открытие пропуска в BLASTP при расчете веса), то остается предположить, что это связано с тем, что BLASTP использует матрицу BLOSUM62, а needle и water - EBLOSUM62. Другие параметры выравнивания в needle объясняются большей длиной выравнивания.
©2008 Михальченко Алексей