Пример использования трехмерного QSAR анализа для предсказания активности низкомолекулярных соединений в отношении данного белка.
1) Предсказание активности ингибитора (подготовка файлов)
Дан набор из 88 веществ – ингибиторов тромбина (compounds.sdf). Для 85 из них активность известна, для трех – предстоит предсказать.
Для начала необходимо построить пространственное выравнивание активных конформаций исследуемых веществ. Будем считать активной конформацией (то есть конформацией, в которой вещество-ингибитор взаимодействует с белком-мишенью) наиболее энергетически выгодную конформацию (часто это вполне соответствует истине). Попробуем сгенерировать эти конформации, используя программу obconformer из пакета OpenBabel:
obconformer 100 100 compounds.sdf > compounds_best_conformer.sdf
Далее необходимо сделать выравнивание полученных конформеров. Попробуем сделать это с помощью программы Open3DALIGN (open3dalign.sourceforge.net).
open3dalign.sh
Чтобы сделать выравнивание нужно загрузить SDF файл со структурами веществ (команда import), выполнить выравнивание (чтобы использовать в качестве темплэйта, к которому выравниваются все вещества, первое вещество в списке, наберите align object_list=1), и записать выравнивание в файл (save).
Перекодировать из юникода в ascii:
iconv -c -f utf-8 -t ascii aligned.sdf > aligned_ascii.sdf
Удалить ненужную информацию из заголовков и добавить $$$$ в конец каждой записи:
sed -e 's/.*HEADER.*\([0-9][0-9]\).*/\1/' -e 's/\(.*M END.*\)/\1\n$$$$/' aligned_ascii.sdf > temp
sed -n '/^[0-9a-zA-Z \$\.-]*$/ p' temp > aligned_ok.sdf
rm temp
2) 3DQSAR анализ
open3dqsar.sh
import type=sdf file=aligned_ok.sdf
import type=dependent file=activity.txt
Активности трех последних соединений нам предстоит предсказать, поэтому для них пока что указана нулевая активность.
Задайте решетку вокруг исследуемых соединений:
box
Давайте оставим часть наших соединений в качестве тестового набора, и не будем использовать их для построения модели, а также исключим (пока что) соединения с неизвестной активностью:
set object_list=60-85 attribute=TEST
set object_list=86-88 attribute=EXCLUDED
Рассчитаем значения энергии ван-дер-Ваальсовых взаимодействий в узлах решетки:
calc_field type=VDW force_field=MMFF94 probe_type=CR
В некоторых узлах решетки псевдо-атом зонда (probe) находится слишком близко к атомам исследуемых содеинений, и дает слишком большую по модулю энергию. Установим ограничения на значения энергии:
cutoff type=max level=5.0 field_list=1
cutoff type=min level=-5.0 field_list=1
Слишком маленькие значения энергии приравняем к 0:
zero type=all level=0.05
Исключим из анализа ячейки, в которых вариабельность в энергии взаимодействия с зондом для разных соединений мала:
sdcut level=0.1
nlevel
remove_x_vars type=nlevel
Построим регрессионную модель:
pls
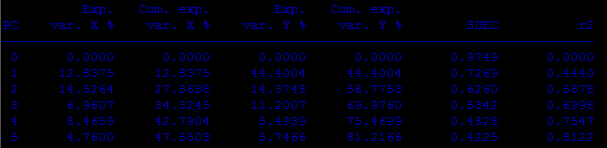
Получаем коэффициенты корреляции (r2) для пяти компонент, выделенных PLS. И они нас устраивают, для 3х компонент они близки к 1.
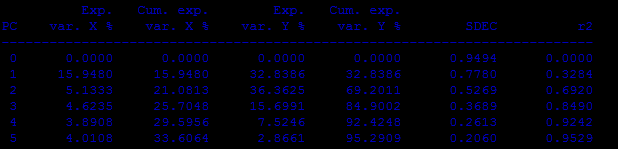
Кросс-валидация:
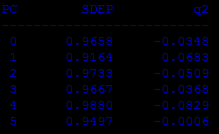
cv type=loo runs=20
Предсказание активности для тестовой выборки:
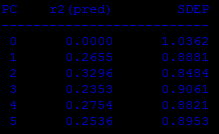
predict
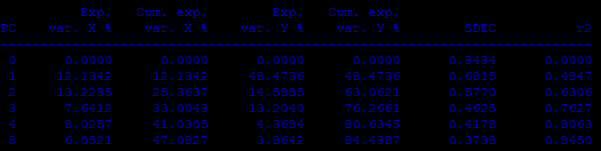
2) Анализ, используя выравнивание и конформации, полученные с учетом структуры активного центра белка-мишени
Регрессионный анализ
Кросс-валидация
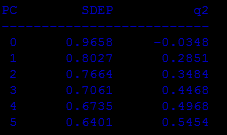
Предсказание активности
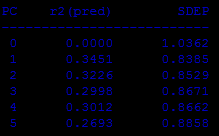
3) Предсказание активности
Для начала, давайте переделаем модель с использованием всех имеющихся данных, а вещества с неизвестной активностью обозначим как тестовую выборку:
set object_list=60-85 attribute=TRAINING
set object_list=86-88 attribute=TEST
Построение модели
Предсказание
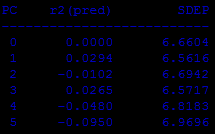

Пусть предсказание будет соответствовать 4ой компоненте (лучше проявила себя в кросс-валидации)
Теперь сделаем модель в Pymol о влиянии заместителей на активность соединений:
© Шерстюк Александра, MSU 2009









