- 3-гидролазы
- 3.5-действует на углерод-азотные связи, кроме пептидных связей
- 3.5.1-в линейных амидах
- 3.5.1.2-глутаминаза
Дополнительная информация:
- IUPAC была организована в 1919 году.
- Международная комиссия по номенклатуре ферментов была организована в 1956 году.
- Последние изменения/уточнения в номенклатуре ферментов были утверждены 31 марта 2006 года. Обновление происходит довольно часто.
Использование ресурса Brenda для характеристики фермента с кодом EC 3.5.1.2
Ферментативная реакция: L–Glutamine + H2O = L–Glutamate + NH3Эта же реакция с использованием структурных формул:
| + | 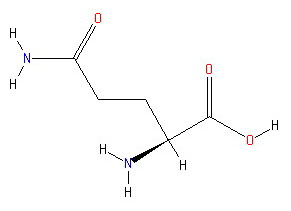 |
= | 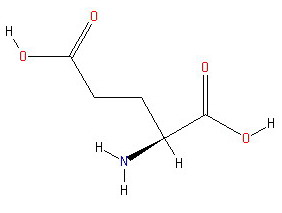 |
+ | 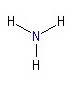 |
С таким же кодом было найдено 104 фермента.
Активаторы:
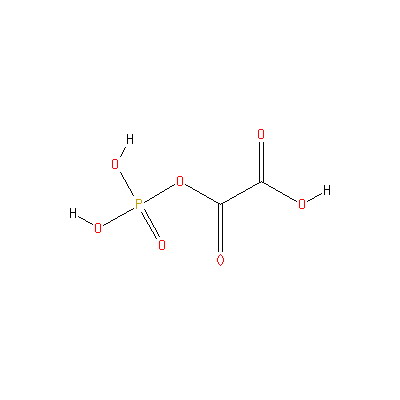 |
|
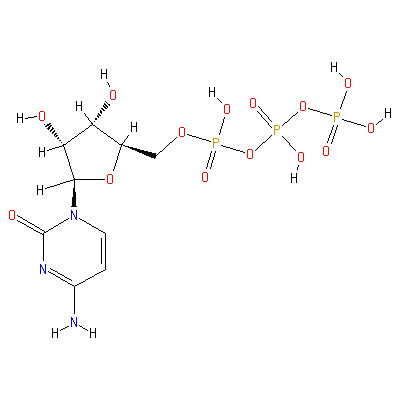 |
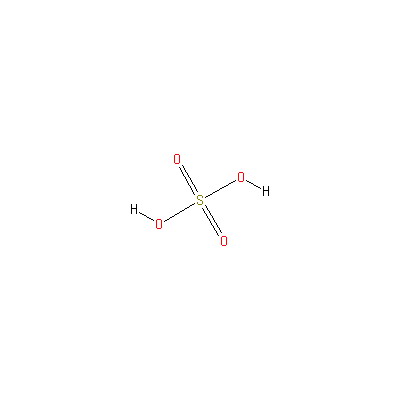 |
Ингибиторы:
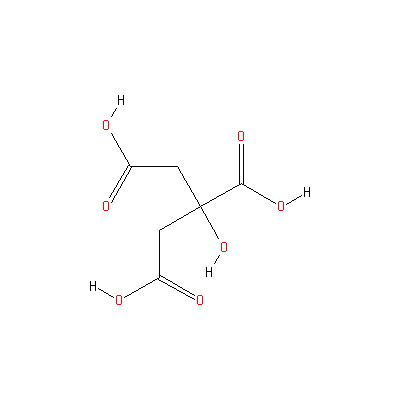 |
|
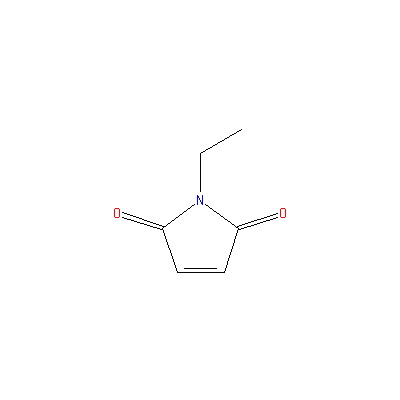 |
 |
Оптимум pH лежит в диапазоне 7-9 в зависимости от организма, например:
- Bacillus pasteurii: 9
- Canis familiaris: 7.9
- Lactobacillus rhamnosus: 7
Температурные оптимумы:
- Bacillus pasteurii: 37
- Micrococcus luteus: 50
- Lactobacillus rhamnosus: 50
Болезни человека, так или иначе связанные с этим ферментом:
-
Alkalosis (Алкалоз). Активация глутаминазы печени является компенсацией при частичном
сокращении синтеза мочевины; одновременно с этим повышается pH крови и увеличивается концентрация бикарбонатов в ней.
- Diabetes Mellitus, Non-Insulin-Dependent
(инсулин-независимый сахарный диабет). Инактивация глутаминазы может играть роль в способности
кишечника переключаться на другие источники питания: с глутамина на кетоновые тела - продукты усиленного
распада клеточного жира - во время диабета и мясного голодания.
- Liver Cirrhosis (цирроз печени).
Глутаминаза и глутамин-синтетаза активируют цирроз печени и гепатоцеллюлярный рак.
- Metabolic acidosis, NOS (метаболический ацидоз). Глутаминаза I системы гипофиз-надпочечники и коры почек является функциональной единицей в гомеостатическом ответе на метаболический ацидоз.
Субъединичная структура:
- Гомодимер: Bacillus pasteurii
- Димер: Micrococcus luteus
- Тример: Rattus norvegicus
- Тетрамер: Rattus norvegicus, Pseudomonas sp., Acinetobacter glutaminasificans
- Более субъединиц: Rattus norvegicus
Пострансляционные модификации: В организме Rattus norvegicus в митохондриях первые 16 аминокислот удаляются.
Сравнение ферментов с одинаковым кодом из эволюционно далеких организмов
По заданному коду EC 3.5.1.2 с помощью SRS в SWISS–Prot искались ферменты по трём хорошо изученным организмам, но было найдено только из двух: из кишечной палочки Escherichia coli K–12 и человека. По архее Methanococcus jannaschii ничего не найдено.Запрос: Query "([swissprot-ECNumber:3.5.1.2] & (([swissprot-ID:*_Ecoli] | [swissprot-ID:*_Metja]) | [swissprot-ID:*_Human])) " found 4 entries
| ID | идентификатор Pfam | положение домена Pfam |
| GLSA1_ECOLI | PF04960 | 24...308 |
| GLSA2_ECOLI | PF04960 | 24...308 |
| GLSK_HUMAN | PF04960 | 244...530 |
| GLSL_HUMAN | PF04960 | 177...463 |
У человека было найдёно несколько доменов, но для сравнения брался только с номером PF04960, потому что он похож на таковой у кишечной палочки. Далее сравнивались попарно домены человека и кишечной палочки. Для получения последовательностей использовалась команда в Linux:
из которых домены были вырезаны с помощью другой команды:
Для получения идентичностей был написан скрипт:
needle GLSA1_ECOLI.txt GLSK_HUMAN.txt -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' > identity.txt needle GLSA1_ECOLI.txt GLSL_HUMAN.txt -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' >> identity.txt needle GLSA2_ECOLI.txt GLSK_HUMAN.txt -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' >> identity.txt needle GLSA2_ECOLI.txt GLSL_HUMAN.txt -gapopen 10 -gapextend 0.5 stdout | grep '# Identity' >> identity.txtДля его запуска использовалась команда
./script
Здесь script — это название файла, в котором записан скрипт. Полученный результат:
# Identity: 111/292 (38.0%) # Identity: 107/296 (36.1%) # Identity: 107/289 (37.0%) # Identity: 98/296 (33.1%)Как видно, сравнивались домены только из эволюционно далёких организмов. Приведённые цифры свидетельствуют о приблизительно одинаковой идентичности — чуть менее 40%. Белки GLSA2_ECOLI и GLSL_HUMAN сравнительно менее похожи, но это незначительно. В целом такая неплохая идентичность может говорить о выполнении этими доменами одной и той же функции.