Занятие 2. Cеми-эмпирические вычисления: Mopac
Ваша рабочая директория: H:\Term6\Practice2. Отчёт по заданию должен появиться на сайте к следующему занятию. Необходимые сведения о работе с Mopac см. здесь. Вся работа по расчётам и конвертированию файлов будет проходить на сервере kodomo через putty. Начнём работу с установки переменных:
Суть задания состоит в поэтапном освоении возможностей Mopac, как пакета для оптимизации структуры молекул и расчёта некоторых свойств.
Прочитайте помощь к программе babel: babel -H. Найдите в интернете порфирин и его аннотацию в виде SMILES (например http://www.ebi.ac.uk/chebi/searchId.do?chebiId=CHEBI:8337). В рабочей директории создайте текстовый файл 1.smi где сначала SMILES порфирина, а через несколько пробелов просто porphyrin.
На основе этого описания c помощью программы из openbabel можно построить 3D структуру порфирина:
1 obgen 1.smi > 1.mol
Просмотрите полученную структуру в PyMol и удалите возможные ненужные водороды, сохраните результат как pdb. С помощью openbabel переформатируйте координаты в mol формате во входной файл для Mopac.
1 babel -ipdb myfile.pdb -omop 1_opt.mop -xk "PM6"
С помощью -xk мы задали тип параметризации pm6.
Запустите Mopac :
1 MOPAC2009.exe 1_opt.mop
Изучите файл вывода out. Для сравнение переформатируйте результат 1_opt.out в pdb:
1 babel -imopout 1_opt.out -opdb 1_opt.pdb
Дополнительное упражение. Проведите оптимизацию с параметризацией AM1 , т.е. :
1 babel -ipdb myfile.pdb -omop 1_opt.mop -xk "AM1"
Сравните структуры полученные obgen и разными способами в Mopac, выводы, картинки и наблюдения занесите в отчёт.
- Рассчитайте возбужденные состояния порфирина и на основе этих данных прикиньте спектр поглощения молекулы. Для расчёта возбуждённых состояний сделайте копию mop файла из предыдущего задания. Например 1_opt_spectr.mop. Для указания Mopac о необходимости расчёта возбуждённого состояния добавьте в конец файла:
cis c.i.=4 meci oldgeo some description
Запустите Mopac:
1 MOPAC2009.exe 1_opt_spectr.mop
Найдите в конце файла значения энергий для электронных переходов. На основании этих значений и простой формулы  рассчитайте длину волн при которых происходят эти переходы. Пара ссылок в помощь.
рассчитайте длину волн при которых происходят эти переходы. Пара ссылок в помощь.
- Для молекулы O=C1C=CC(=O)C=C1 определите геометрию как с помощью obgen так и Мopac (основные шаги см. выше). Определите геометрию дианиона этой молекулы. Для начала в первую строчку mop файла добавьте слово CHARGE=-2. Потом явным способом укажите на каких атомов по вашему мнению должен находиться отрицательный заряд. Пример:
PM6 CHARGE=-2
gg
O(-) 0.98570 1 0.00130 1 -0.43680 1
C 2.16830 1 0.00680 1 -0.12400 1
...........Традиционно сравните молекулы. Рекомендую ball & stick отображение. Запишите ваши наблюдения и обсуждение в отчёт.
Известно, что ультрафиолет может превращать тимины в тиминовые димеры, так же известно, что ДНК фотолиаза при облучении ультрафиолетом востановливает основания тиминов до нормального. Вам дан тиминовый димер
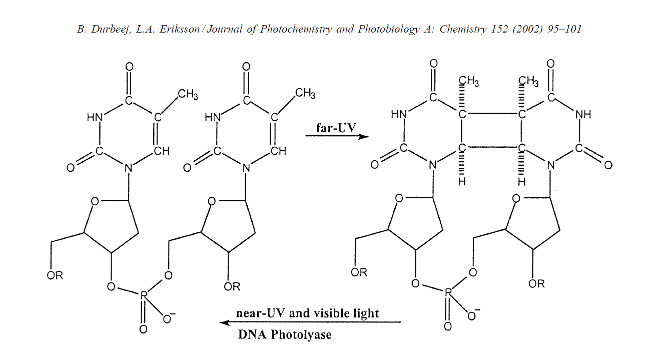
Наша цель увидеть переход из димера в тимины при возбуждении системы. Так как вычесления вобуждённых состояний в MOPAC затруденены, мы имитируем возбуждение ионизируя оба кольца, т.е. указывая заряд системы +2. И полученое возбуждённое состояние снова оптимизируем при заряде 0.
- Проведите оптимизацию геометрии этого димера, при заряде системы 0.
- Проведите оптимизацию результата из пункта i. , при заряде системы +2.
- Проведите оптимизацию результата из пункта ii. , при заряде системы 0.
Сравните энерегии всех состояний. Опишите причину по которой по Вашему мнению возбуждённое состояние при добавлении электронов не перешло обратно в исходно состояние.
Последнее задание самое сложное и поэтому дополнительное. Вам дана некоторая конформация где АТФ связывается с белком через координацию иона магния, но магния в самой структуре нет. Вам надо определить координацию иона Mg молекулами воды, белком и гамма-фосфатом.
- Сначала надо добавить водороды, но для фосфатной группы важен рН среды. Эту операцию можно сделать с помощью babel.
- Добавьте вручную в файл атом магния где-то меду гамма-фосфором и СА аспартата. Можно просто скопировать атом фосфора и поменять имя атома на Mg и поменять координаты как среднее арифметическое между координатами гамма-фосфора и СА аспартата.
- Переведите полученные pdb координаты в форматы mop и xyz.
- Следующим этапом будет указание запрета на движение для всех атомов кроме гамма фосфата, воды и магния. Этот шаг нам нужен для того, чтобы потом легко восстановить эту конформацию в белке. Для "заморозки атомов" надо в mop файле поменять 1 после координаты на 0. Пример:
PM6 CHARGE=-3
test
N -33.26800 0 25.03000 0 4.67600 0
.......Как удобно определить каким атомам надо разрешить двигаться? Откройте xyz файл в PyMol и отобразите labels как atoms id. Номер строчки с этим атомом в mop файле будет id+3. Таким образом установите у нужных атомов 1 по всем координатам.
- Проведите оптимизацию и сравните со стартовой конформацией. Опишите изменения. Опишите координацию магния. Сравните результат с pdb записью 3pp1.


 2023
2023 2022
2022 2021
2021 2020
2020 2019
2019 2018
2018
 2017
2017 2016
2016